10月3日,國際學術期刊Nucleic Acids Research在線發表了中國科學院上海營養與健康研究所生物醫學大數據中心張國慶研究員與中國科學院分子植物科學卓越創新中心周志華研究組的合作論文“RDBSB: a database for catalytic bioparts with experimental evidence”。該研究構建了首個面向合成生物學設計的催化元件數據庫RDBSB,提供了83,193個具有實驗證據的催化元件在線查詢平臺(https://www.biosino.org/rdbsb),同時還提供了PathFinder工具,可以根據底物和目標產物設計相應的合成途徑,并能完整復現例如晚期肝細胞癌的治療藥物淫羊藿素和棉鈴蟲性信息素的人工合成途徑設計過程。
催化元件是設計、構建和優化特定代謝途徑乃至生命系統的基礎,一個可靠的、經過實驗驗證的催化元件數據庫對于設計和優化這些生命系統至關重要。而現有的生物元件庫缺乏底盤、催化反應最佳pH和溫度等對合成過程至關重要的信息。為了填補這一空白,研究團隊從各種數據庫整合了總計390,708個催化元件,通過審編后得到了83,193個有實驗驗證的催化元件,包含大量的轉移酶、水解酶和氧化還原酶(圖1A)。其中,3200個催化元件包括最適催化反應溫度(20°C至40°C)、最適pH值(6-9)等關鍵信息(圖1B)。這些催化元件大部分來源于細菌、后生動物和植物(圖1C)。
基于此,研究團隊設計并構建了合成生物學元件和數據庫(以下簡稱RDBSB),在線提供系統審編的定性和定量催化信息,包括元件的活性、底物、催化反應最佳pH值和溫度以及底盤特異性等關鍵參數。平臺還提供了包括MapView、AlphaFold、PVQD和PathFinder等在內的豐富的在線元件和途徑設計工具。通過RDBSB,用戶不僅可以在線搜索并查閱催化元件詳細的定性和定量信息及其對應的參考文獻,還可以提交新的催化元件,促進數據的快速共享與利用。截至目前,RDBSB已為一千多個催化元件共享提供了支撐。該數據庫將極大豐富合成生物學途徑設計的可用資源,為研究人員提供必要的工具。
中國科學院上海營養與健康研究所生物醫學大數據中心高級工程師劉婉、博士研究生莊心昊、中國科學院分子植物科學卓越創新中心副研究員王平平為論文共同第一作者。中國科學院上海營養與健康研究所張國慶研究員和中國科學院分子植物科學卓越創新中心周志華研究員、嚴興研究員為論文共同通訊作者。該研究得到了科技部國家重點研發計劃、中國科學院戰略性生物資源服務網絡計劃等項目的支持。
文章鏈接:https://academic.oup.com/nar/article-lookup/doi/10.1093/nar/gkae844
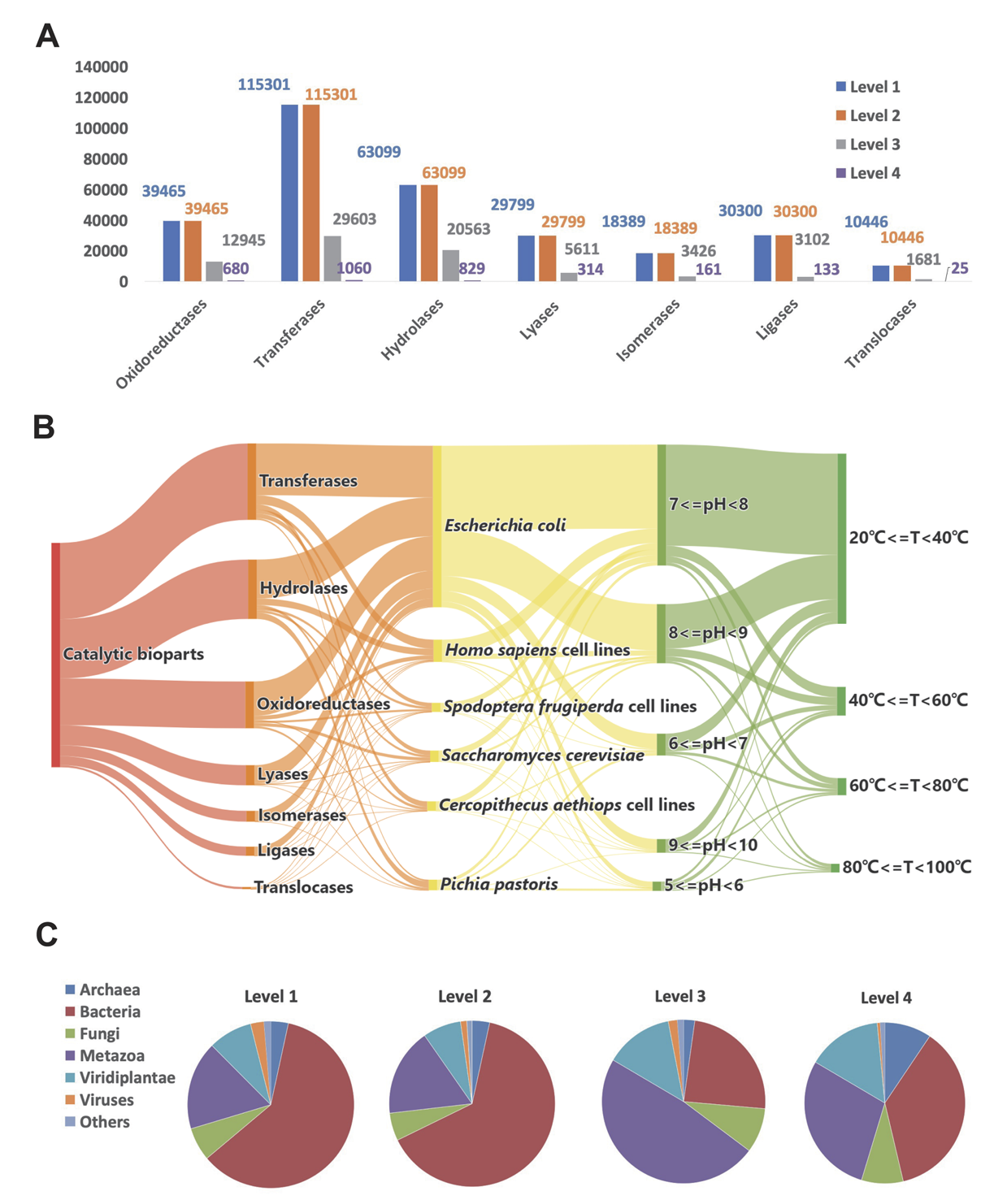
圖1:RDBSB數據概況。(A)催化元件在ENZYME數據庫分類的分布情況。(B)經實驗驗證的催化元件在酶類別、底盤、最佳pH值和最佳溫度的分布。(C)根據NCBI分類法分類的元件來源生物的分布情況。
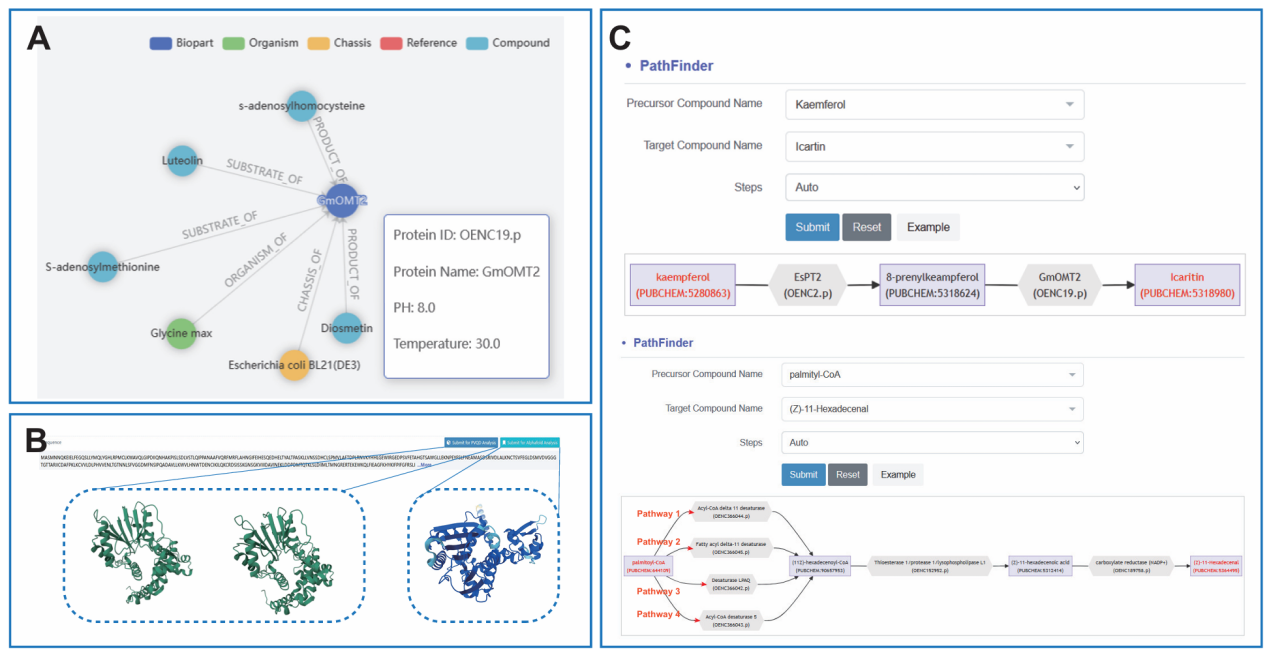
圖2:RDBSB在線分析和設計功能。(A)GmOMT2的MapView示例。視圖提供催化元件、底盤、物種和化合物之間的關系。(B)利用PVQD和AlphaFold對GmOMT2進行三維構象和結構預測。(C)PathFinder復現人工生物合成途徑設計的實例:酵母中晚期肝細胞癌治療藥物淫羊藿素的人工合成途徑和棉鈴蟲性信息素的人工合成途徑。